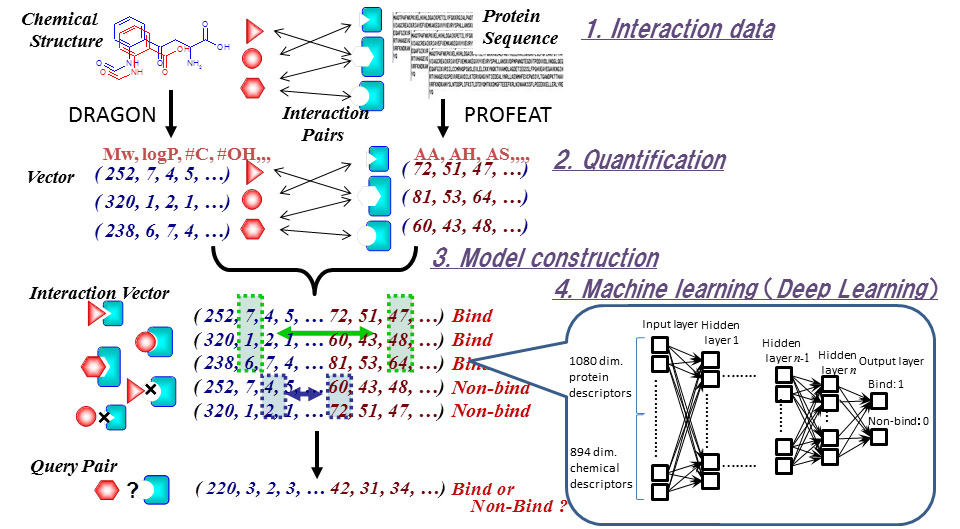
本稿では,医薬品となる化合物を発見するための第一段階のスクリーニングである,タンパク質と化合物の相互作用の予測について述べる.膨大な種類の化合物から医薬品になり得るリガンド化合物を見つけ出す工程は,開発にかかる時間とコストを押し上げる主要因となっている.我々はこれまで,相互作用が確認された12.5万件の結合データと,結合データに含まれない同数の組み合わせを非結合データとして用意し,それらをサポートベクターマシンで学習することで相互作用を予測するCGVBS法を提案してきたが,データが増えるにつれて学習時間が長大になることや,学習データが少数追加された場合でも再度学習をやりなおさなくてはならないなど,今後大規模な相互作用データを学習していく上で検討すべき課題があった.そこで,相互作用予測にDeep Learning(深層学習)の一手法である,Deep Belief Networks(DBN)を用いたCGBVS-DBN法を提案する.400万件のデータセットを用いた評価実験の結果,CGBVS-DBN法は98.2%の精度で相互作用が予測可能であった。
図:
論文
- [LINK] [創薬] 浜中雅俊: “in silico創薬におけるスクリーニングの高速化・高精度化技術”より, “第2章 スーパーコンピュータ・人工知能による創薬・育薬の高速化と最適化”, “10節ディープラーニングを用いた化合物—タンパク質の相互作用予測”, 技術情報協会, 2018.
- [LINK] [創薬] Masatoshi HAMANAKA, Kei Taneishi, Hiroaki Iwata, Jun Ye, Jianguo Pei, Jinlong Hou, Yasushi Okuno: “CGBVS-DNN: Prediction of Compound-protein Interactions Based on Deep Learning”, Molecular Informatics, Vol.36, Issue1-2, Wiley, 2016.
- [PDF] [創薬] Masatoshi Hamanaka, Kei Taneishi, Hiroaki Iwata, Yasushi Okuno: “Prediction of Compound-Protein Interactions based on Deep Learning”, Proceeding of The 6th French-Japanese Workshop on Computational Methods in Chemistry, March 2016.
- [LINK] [創薬] Masatoshi Hamanaka, Kei Taneishi, J.B. Brown, Yasushi Okuno: “Prediction of compound-protein interactions based on deep-layered learning”, The International Chemical Congress of Pacific Basin Societies 2015 (PACHIFICHEM), December 2015.
- [PDF] [創薬] 浜中雅俊, 種石慶, 岩田浩明, 奥野恭史: “ディープラーニングに基づく化合物とタンパク質の相互作用予測”, 第38回ケモインフォマティクス討論会, O10, pp. 46-49, October 2015.
- [PDF] [創薬] 浜中雅俊, 種石慶, J.B.Brown, 奥野恭史: “深層学習に基づくタンパク質と化合物の相互作用予測”, 第77回 情報処理学会全国大会 大会優秀賞Best Paper Award of IPSJ National Convention, 2015.